Warum das wichtig ist: Die weithin bekannte Genschere CRISPR/Cas9 entstand nicht über Nacht. Schon vor der bahnbrechenden Publikation 2012 in der Fachzeitschrift „Science“, die CRISPR/Cas als Geneditierungs-Technik beschrieb, konnten Forscher:innen die DNA gezielt verändern. Heute spricht kaum mehr jemand über Zinkfingernukleasen oder TALENs, dabei wurden sie schon vor CRISPR/Cas9 zur Genom-Editierung angewendet – sie sind sozusagen die älteren Geschwister.
„Genom-editierte Embryonen in China.“ Das war wohl die Wissenschaftsschlagzeile im Jahr 2018. Doch schon in den Jahren zuvor meldeten die Medien in regelmäßigen Abständen von neuen Durchbrüchen in der Biologie. Viele dieser bahnbrechenden Entdeckungen wurden erst durch die Anwendung eines Systems namens CRISPR/Cas9 ermöglicht. Die Anwendung dieses bakteriellen Immunsytems zur gezielten Gen-Veränderung wurde erstmals 2012 publiziert (Jinek et al, 2012). Auch die Anzahl der wissenschaftlichen Publikationen zum Stichwort „Gen-Editierung“ steigt seit 2012 in ähnlichem Maß wie die Anzahl der Publikationen zum Stichwort „CRISPR/Cas9“ (PubMed - NCBI). Eine besondere Verbindung hat die Genschere zu Österreich: Eine der Entdecker:innen dieses revolutionären Tools, Emmanuelle Charpentier, war bis 2019 im Max F. Perutz Labor in Wien tätig.
Die Möglichkeit einer gezielten Veränderung von Genen wurde durch CRISPR/Cas9 einer breiten Öffentlichkeit ins Bewusstsein gerufen. Die Debatten, wo die Grenzen dieser Eingriffe zu ziehen sind, werfen rechtliche, ethische und gesellschaftliche Fragen auf. Doch Wissenschaftler:innen können nicht erst seit der erstmaligen Publikation dieses Genomveränderungs-Tool in das Erbgut eingreifen.
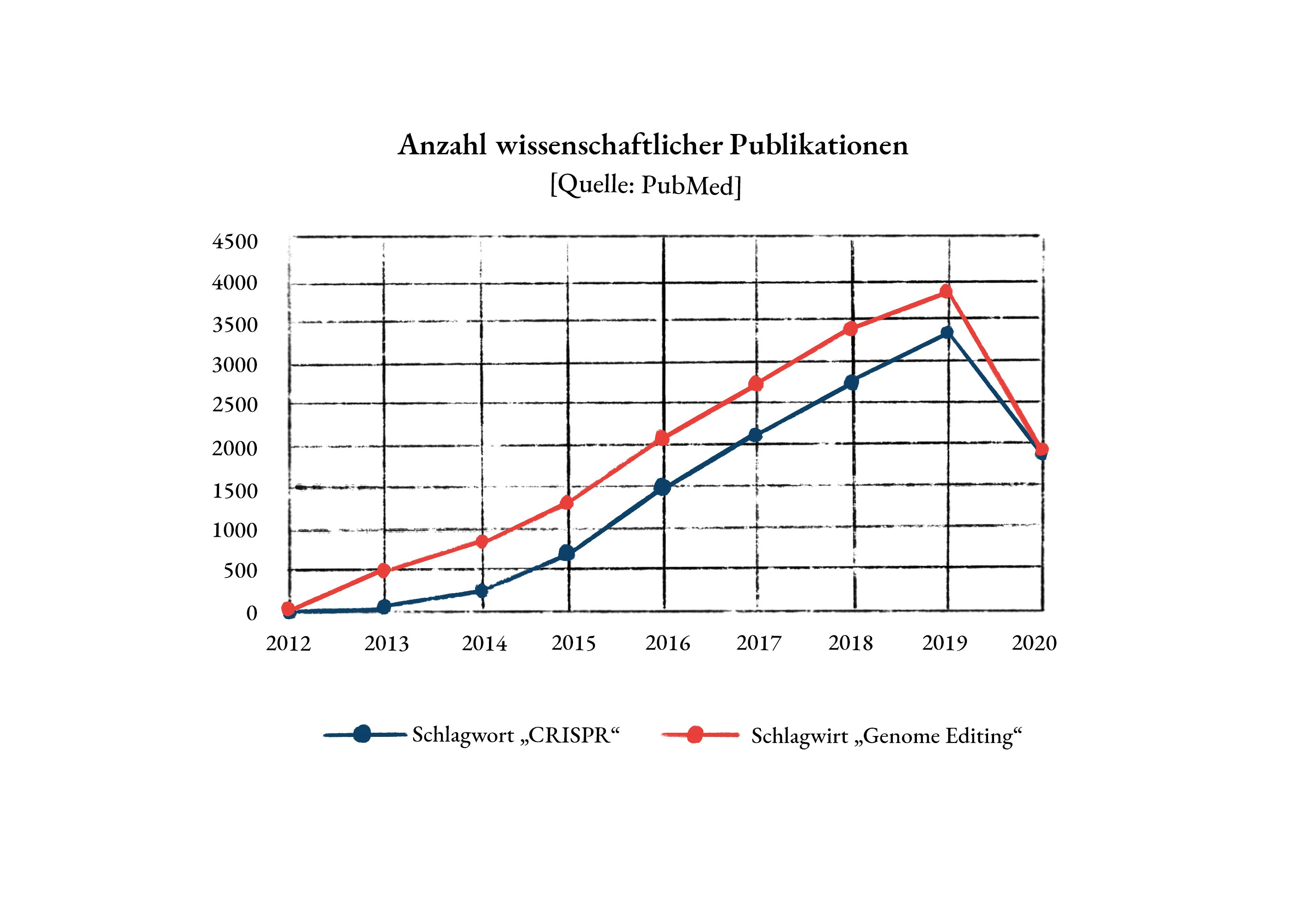
Abbildung 1: Die Publikationen rund um CRISPR/Cas9 stiegen in den letzten Jahren stark an.
(Graphik: Carmen Tung)
Der Wunsch nach Veränderung der Erbinformation
Die Erbinformation (DNA) unterliegt einem ständigen Wandel durch natürliche Prozesse. Beispielsweise ruft UV-Strahlung Veränderungen, sogenannte Mutationen, in der DNA hervor. (Schmidt, 2017)
Seit Anbeginn der Menschheit besteht ein Wunsch nach gezielter Veränderung der DNA. Doch vor rund 13. 000 Jahren ging es nicht um die Verbesserung des Menschen. Am Anfang stand die Notwendigkeit möglichst ertragreiche Pflanzen anzubauen. (Brown, 2011 / Heberer, 2015)
Die Menschen erkannten schon sehr bald, dass dies durch Kreuzen von Pflanzen mit vorteilhaften Eigenschaften erreicht werden konnte. So veränderten schon unsere Vorfahren die DNA von Nutzpflanzen durch Versuch und Irrtum. (Kempken & Kempken, 2012)
Je mehr Erkenntnisse die Forschung über die Erbinformation, den Aufbau der DNA und deren Funktionen brachte, desto größer wurde der Wunsch nach gezielten Veränderungen. Angetrieben wurde die Forschung auch durch die Erkenntnisse, wie viele Erkrankungen eine genetische Komponente haben. In der gezielten Veränderung der DNA hoffte man somit neue Behandlungsmöglichkeit zu finden.
Molekularbiologische Methode als Schlüssel
Den ersten Schritt des Weges zur gezielten Veränderung der DNA machte die Erkenntnis, dass die Erbinformation als Doppelhelix aufgebaut ist. Hierbei werden je zwei der Bausteine der DNA, sogenannter Nukleotide, über spezielle Bindungen, Wasserstoffbrückenbindungen, zusammengehalten. Die Paarungen sind dabei aufgrund der chemischen und strukturellen Beschaffenheit fix vorgegeben: Adenin (A) mit Thymin (T) und Guanin (G) mit Cytosin (C). (Watson & Crick, 1953)
Die ersten Werkzeuge der Molekularbiologie, die schließlich ein gezieltes Eingreifen in die DNA ermöglichten, waren die sogenannten Restriktionsendonukleasen, allgemein als Restriktionsenzyme bezeichnet. Ende der 1960er-Jahre wurden diese Enzyme von den späteren Nobelpreisträgern Werner Arber, Daniel Nathans und Hamilton O. Smith entdeckt. Da sie spezifische Sequenzen erkennen und an diesen Stellen die DNA schneiden, werden sie auch als „Molekulare Scheren“ bezeichnet. Es handelt sich dabei um natürlich in Bakterien vorkommende Enzyme, die von den Bakterien als Immunsystem gegen Eindringlinge eingesetzt werden. (Graw, 2015)
Sie machten die meisten molekularbiologischen Versuche erst möglich. Jedoch kann mit Restriktionsenzymen nicht jeder beliebige Abschnitt der DNA verändert werden. Es muss eine spezielle Sequenz in der DNA vorhanden sein, die dann vom Enzym erkannt und in einem bestimmten Bereich geschnitten wird. Meist ist diese bestimmte Abfolge von A, T, G und C ein Palindrom in der Länge von 4, 6 oder 8 Basenpaaren. Das bedeutet, dass sich die Abfolge der Basen auf den beiden DNA-Strängen spiegeln. Wenn also auf dem einen Strang von links nach rechts gelesen die Sequenz CCCGGG lautet, so ist aufgrund der festgelegten Paarungen der Nukleotide die Sequenz am gegenläufigen Strang GGGCCC. Diese Erkennungssequenzen sind spezifisch für ein Enzym. Das heißt, wenn andere DNA-Abschnitte geschnitten werden sollen, muss dafür ein anderes Enzym verwendet werden. (Mülhart, 2013)
Da in weiten Teilen des Genoms überhaupt keine Erkennungssequenz für Restriktionsenzyme vorkommen, sind gezielte Veränderungen und Eingriffe in das gesamte Genom eines Organismus, die Gesamtheit aller Gene, schwierig bis unmöglich. Ab dem Zeitpunkt, an dem es möglich wurde, mit neuen Techniken gezielt Gene eines Organismus zu verändern, spricht man von Genom-Editierung.
Natürliche Mechanismen nutzen
Ein weiterer wichtiger Aspekt von Genom-Editierungsmethoden ist die Nutzung von natürlichen, zellulären Mechanismen.
Der verursachte Schnitt in der DNA wird wissenschaftlich als Doppelstrangbruch (DSB) bezeichnet. Solche Doppelstrangbrüche entstehen auch durch natürliche Vorgänge, beispielweise durch UV-Strahlung. Um den Verlust von Erbinformation zu verhindern, muss so ein Bruch repariert werden. Hierzu verfügen Zellen über zwei verschiedene Mechanismen, die Homologe Rekombination (HDR - Homology-directed repair) und Nicht-homologe Endverknüpfung (NHEJ – Non-homologous end-joining). (Schmidt, 2017) „Homolog“ kommt aus dem Griechischen und bedeutet „übereinstimmen“.
Bei der homologen Rekombination werden DNA-Enden verknüpft, die aufgrund ihrer Sequenz, der Abfolge der Nukleotide, zusammenpassen. Bei diesem Mechanismus kann auch ein Teil eines intakten DNA-Strangs als Vorlage für die Reparatur dienen, wenn dieser vorhanden ist. (Folger et al, 1982)
Im Gegensatz dazu werden bei der Nicht-homologen Endverknüpfung DNA-Stücke zusammengefügt, die keine Übereinstimmung in ihrer Sequenz haben. Dadurch ist dieser Mechanismus fehleranfälliger als die homologe Rekombination, es kommt vermehrt zu Mutationen. Der Vorteil hierbei ist, dass die Reparaturen meist schneller ablaufen. Das ist für manche Vorgänge wichtiger als eine hohe Präzision, da viele zufällige Mutationen keinerlei Auswirkungen auf den Organismus haben. (Pfeiffer et al, 1994)
Bei Genom-Editierungstechniken werden beide Reparaturmechanismen genutzt. Je nachdem welche Art der Veränderung man hervorrufen möchte, ist der eine oder der andere Mechanismus zu favorisieren. (Voytas & Gao, 2017)
Zinkfingernukleasen bringen die Veränderung
Um das gesamte Genom eines Organismus zu verändern, braucht man zuerst Nukleasen, die in der DNA Doppelstrangbrüche einführen, wie es auch die Restriktionsenzyme tun.
Zusätzlich muss man, im Unterschied zur Anwendung von Restriktionsenzymen, in der Lage sein, den Ort dieses Strangbruchs festzulegen. Dazu braucht es die Möglichkeit, spezifische Erkennungssequenzen bestimmen und je nach Bedarf verändern zu können. Nur wenn diese Voraussetzungen erfüllt sind, kann eine gezielte Veränderung jedes Gens vorgenommen werden.
Als eines der ersten Systeme, das diese Anforderungen erfüllt, wurde Anfang der 90er-Jahre die Kombination von Zinkfingerproteinen mit der Fok I Endonuklease entdeckt. Diese Proteine bestehen aus einer speziellen Domäne, der Zinkfingerdomäne, mit der sie DNA binden können. Ihren Namen verdanken sie ihrer schleifenartigen Struktur und dem daran gebundenen Zinkion. Ein Zinkfinger erkennt immer ein Triplett von Nukleotiden, also drei nebeneinanderliegende DNA-Bausteine. Das künstliche Aneinanderreihen von „Zinkfingern“ ermöglicht das Erkennen eines bestimmten DNA-Abschnittes.
In Kombination mit dem „schneidenden“ Teil eines Restriktionsenzyms, kann so gezielt DNA geschnitten und wieder repariert werden. Bei den Zinkfingernukleasen wird dafür meist die Fok I Endonuklease verwendet, die natürlich im Bakterium Flavobacterium okeanokoites vorkommt. (Li et al, 1992 / Kim et al, 1996)
Die Verbindung von Zinkfingerdomäne und Endonuklease wird als Zinkfingernuklease (ZFN) bezeichnet. Anfang der 2000er-Jahre wurden die ZFNs zur Veränderung des Genoms von pflanzlichen (Ackerschmalwand - Arabidopsis thaliana) und tierischen (Fruchtfliege - Drosophila melanogster) Modellsystemen eingesetzt. (Lloyd et al, 2005 / Bibikova et al, 2002)
Die Herstellung solcher spezifischen Zinkfingernukleasen erfordert viel Erfahrung und ist sehr aufwendig. Da für jede neue Ziel-DNA neue Zinkfingernukleasen generiert werden müssen, ist dies auch sehr kostspielig. (Gaj et al, 2013)
TALEN als neue Hoffnung der Molekularbiologie
Forscher:innen verfolgten nach der Entdeckung der ZFNs als Genom-editierendes Tool die Idee, andere DNA-erkennende Proteine genauso einzusetzen. 2009 stießen sie auf die TALE (Transkription Aktivator-artigen Effektor) Proteine.
In der Natur kommen diese Proteine in den pflanzenbefallenden Bakterien Xanthomonas vor. Wie die Zinkfingerproteine beinhalten sie eine DNA-bindende Domäne. Diese besteht hier aus 33-35 Wiederholungen von Aminosäuren - den Bausteinen von Proteinen. Die meisten der Aminosäuren in Abschnitten von Wiederholungen sind fix definiert. Nur zwei Positionen (12/13) sind variabel und bestimmen so die DNA-Bindestelle. (Moscou et al, 2009 / Boch et al, 2009) Durch die gezielte Veränderung der Aminosäuren an diesen Positionen kann eine definierte Ziel-DNA erkannt werden. Mittels der Kombination mit der Fok I Nuklease, derselben Endonuklease, die auch schon bei den ZFN eingesetzt wurde, kann die DNA an der festgelegten Stelle „geschnitten“ werden. (Christian et al, 2010)
Dieses weitere Mitglied in der Familie der Genom-Editierungsmethoden wird TALEN (Transkription Aktivator-artigen Effektor-Nukleasen) genannt.
Im Gegensatz zu den Zinkfingernukleasen sind die TALENs viel genauer. Allerdings ist auch die Herstellung von spezifischen TALENs sehr kompliziert und teuer. (Gaj et al, 2013)
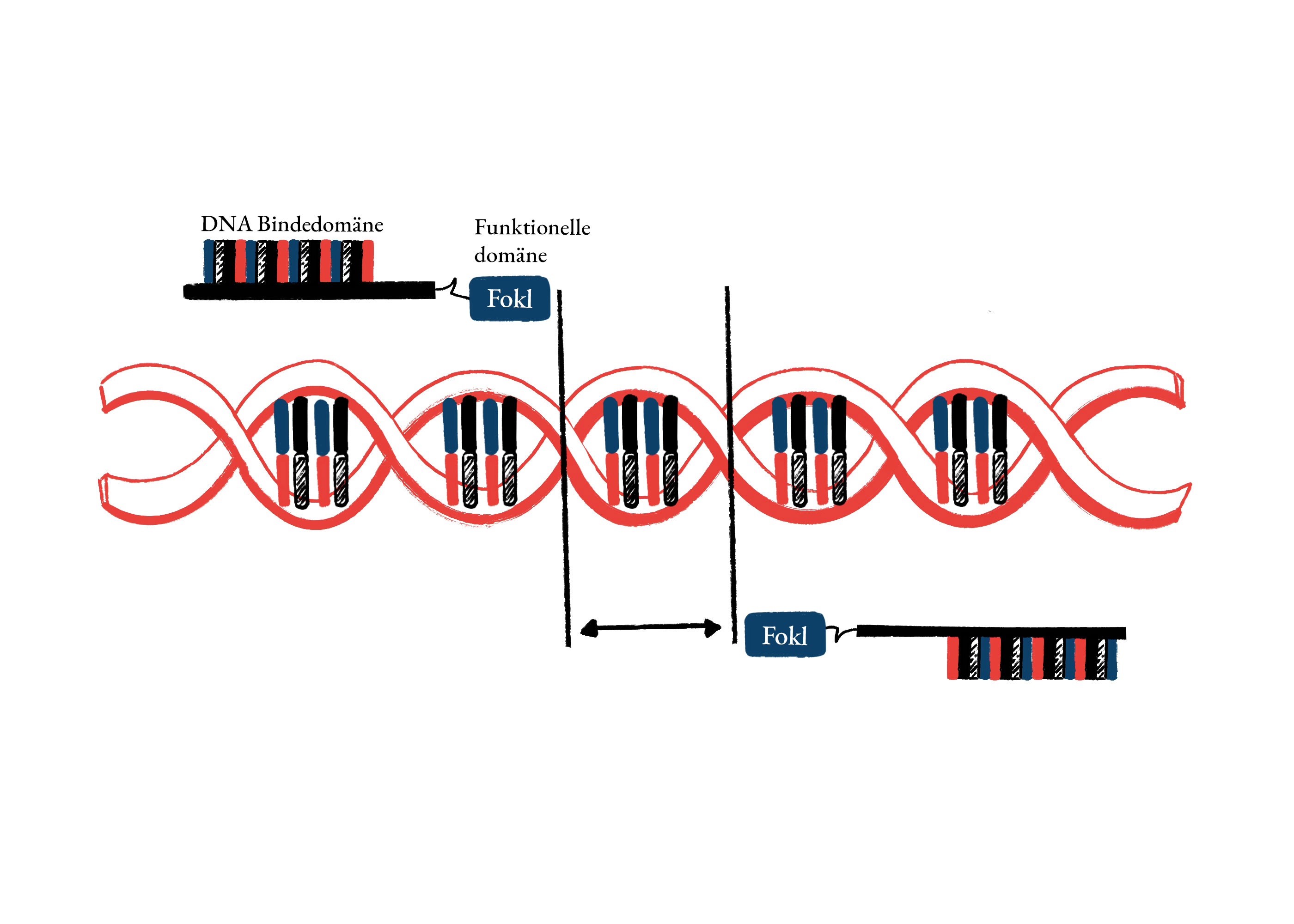
Abbildung 2: Funkionsweise des TALEN-Systems
(Garphik: Garmen Tung; Quelle: Autorin)
Revolution durch ein bakterielles Immunsystem
Die vielgefeierte „Genschere“ CRISPR/Cas9 war also nicht das erste Tool mit dem präzise Eingriffe in das Genom möglich wurden. Auch das System an sich war Forschern schon vor der richtungsweisenden Publikation 2012 bekannt.
Die kurzen, sich wiederholenden DNA-Sequenzen, die als „clustered regularly interspacedshort palindromic repeats” (CRISPR) bezeichnet werden, wurden bereits in den 1980er-Jahren in Japan entdeckt. (Ishino et al, 1987)
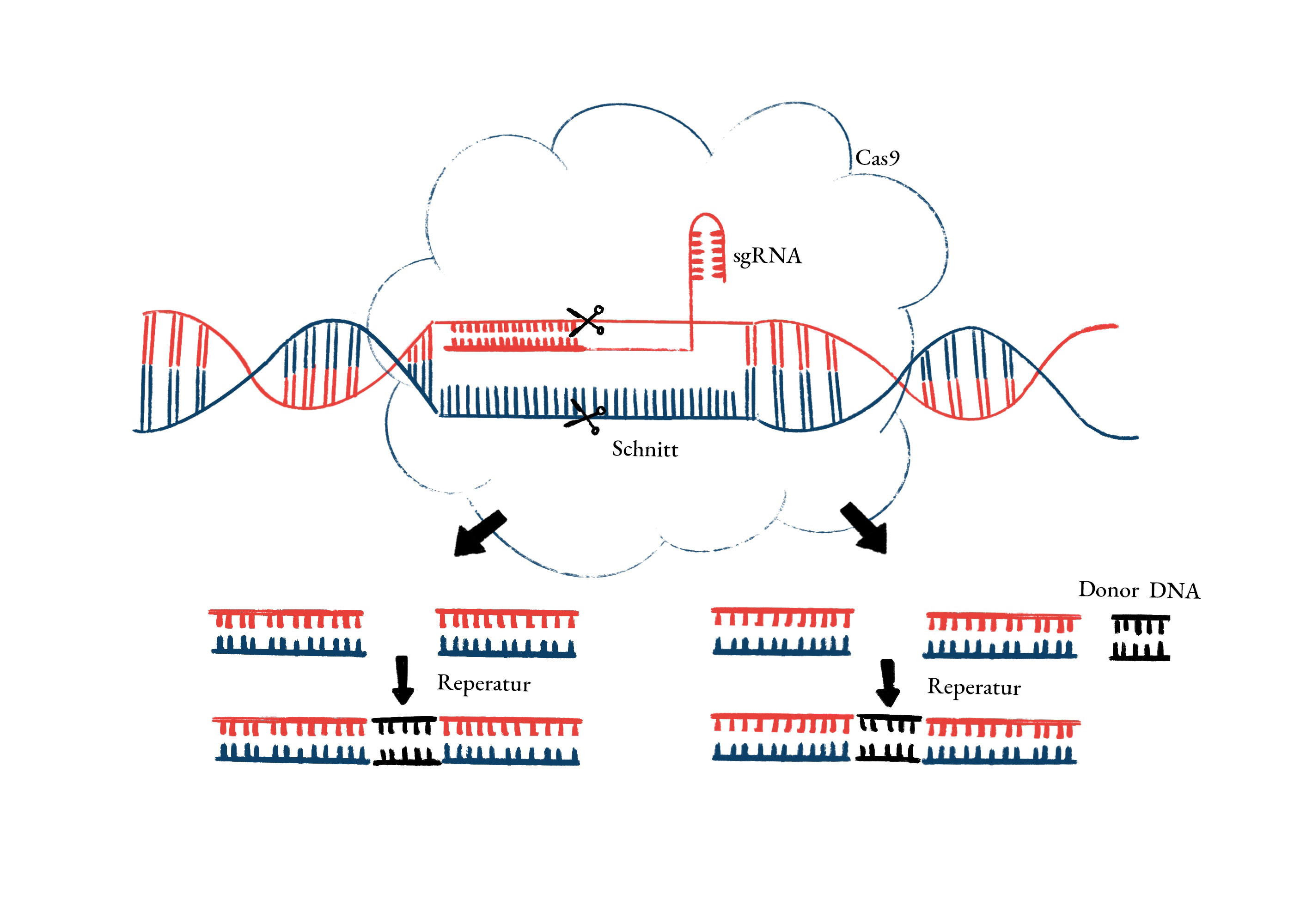
Zusammen mit der Endonuklease Cas9 bilden die CRISPR eine Art “Immunsystem“ in Bakterien. Wird das Bakterium von einem Virus befallen, werden kurze Stücke der viralen DNA zwischen die sich wiederholenden Sequenzen eingefügt und auf diese Weise „abgespeichert“. Bei neuerlichem Befall kann der Eindringling schnell identifiziert werden. Das „schneidende“ Enzym Cas9 wird dazu mittels zweier RNA-Molekülen an die Stelle des viralen Genoms geführt, dessen Sequenz in der CRISPR Abfolge „gespeichert“ wurde. Dort ruft es wie die Fok I Endonuklease bei den anderen Genom-editierenden Verfahren einen Doppelstrangbruch hervor. RNA ist in ihrem Aufbau der DNA ähnlich, allerdings liegt sie nur einzelsträngig vor und wird deshalb zum Transfer von genetischer Information genutzt. (Jinek et al, 2012)
Für das revolutionäre Tool, in dieser Form erstmals 2012 beschrieben, wurden die beiden unabhängigen RNA-Moleküle zu einer einzigen, sogenannten Leit-RNA (sgRNA) zusammengefügt. Diese führt dann genau wie im natürlichen System die Cas9-Endonuklease an jene Stelle, die „geschnitten“ werden soll. Die Herstellung und Veränderung von RNAs ist im Vergleich zu den anderen Verfahren relativ einfach und günstig, da nur einzelne Nukleotide ausgetauscht werden müssen, um jede Sequenz im Genom eines Organismus ansteuern zu können. (Jinek et al, 2012)
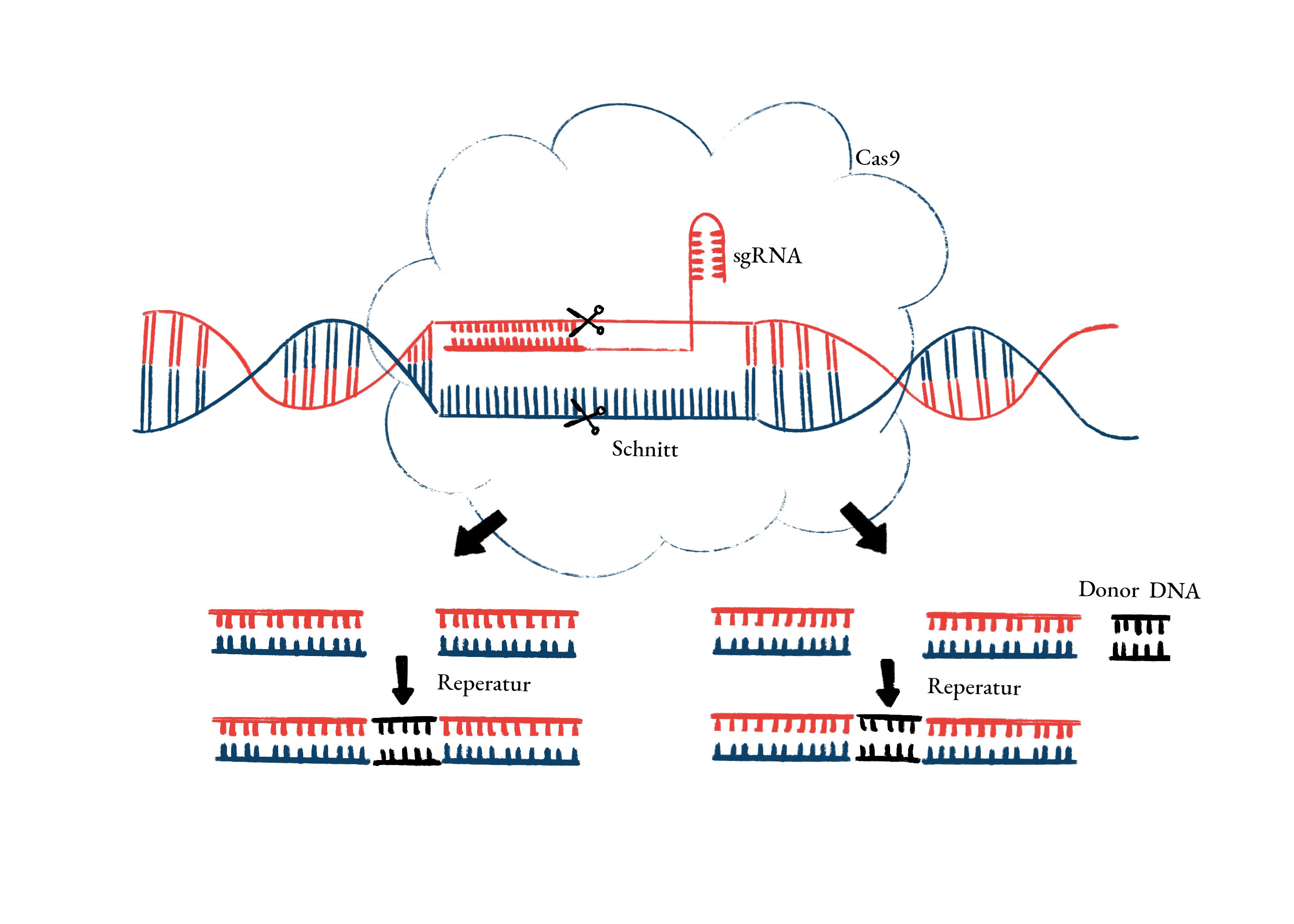
Abbildung 3: Schematische Darstellung CRISPR/Cas9
(Graphik: Carmen Tung; Quelle: Autorin)
Präzision als Familienproblem
Sowohl ZFN als auch TALEN sind nicht nur teuer und kompliziert in der Herstellung, auch ihre Genauigkeit kann ein Problem sein. TALENs sind in diesem Punkt den ZNF überlegen. Das bedeutet: Das fälschliche, unbeabsichtigte Schneiden einer DNA-Sequenz, als Off-Target-Effekt bezeichnet, ist ein Problem, das alle beschriebenen Techniken teilen. (Pattanayak et al, 2014)
Bei allen Geneditierungstechniken werden DNA-Sequenzen von etwa 20 Nukleotiden zur Erkennung verwendet. Rechnerisch liegt die Wahrscheinlichkeit, dass genau diese Sequenz an einer anderen Stelle als der gewünschten noch einmal im menschlichen Genom vorkommt bei 1 zu einer Billion. Das menschliche Genom besteht allerdings aus rund 3 Milliarden Basenpaaren. Es erscheint also unwahrscheinlich, dass es zu einer Veränderung in einem ungewünschten DNA-Abschnitt kommt.
Die Rechnung ist in der Realität aber nicht so einfach. Im Genom gibt es viele repetitive, also sich wiederholende Sequenzen. Hinzu kommen jene Bereiche, die den Bauplan für Proteine enthalten und somit festgelegt sind. Außerdem muss zur Sequenzerkennung keine perfekte Übereinstimmung des DNA-erkennenden Anteils und der Ziel-DNA vorliegen. So kommt es weitaus häufiger zu unerwünschten „Schnitten“ – Off-Target Effekten - als man vermuten könnte. (Rembold, 2017)
Diese Off-Target-Effekte sind auch die größten Probleme, mit der die viel gefeierte Genschere CRISPR/Cas9 zu kämpfen hat. Denn obwohl es sich hierbei um die billigste und flexibelste Methode von allen handelt, müssen noch immer einige Herausforderungen gemeistert werden. Mit Hochdruck und verschiedensten Ansätzen versuchen Wissenschaftler:innen die Genauigkeit weiter zu erhöhen. (März, 2017)
Fest steht allerdings, dass das jüngste Mitglied der Genom-Editierungsfamilie seine älteren Geschwister in allen Eigenschaften, etwa der flexiblen Anpassung, der kostengünstigen Herstellung, der einfachen Handhabung und nicht zuletzt der öffentlichen Popularität, übertrifft.
Dr. Victoria Lunz hat an den Universitäten Salzburg und Linz Molekularbiologie studiert. Für ihre Master- und Doktorarbeit forschte sie an einer speziellen Klasse von Ionenkanälen, den Kalzium-aktivierten-Kalzium Kanälen. Bei ihrer Arbeit kam sie immer wieder in Kontakt mit Zellsystemen, die durch genetische Methoden wie CRISPR/Cas9 verändert wurden. In ihrer Masterarbeit des Studiums Recht und Wirtschaft für Techniker:innen an der Johannes Kepler Universität Linz widmete sie sich den rechtlichen Rahmenbedungen für die Anwendung von Systemen wie CRISPR/Cas9.
- In diesem Video siehst du, wie CRISPR/Cas9 funktioniert: "Gen-editing mit CRISPR/Cas9" (MaxPlanckSociety)
- alexandria hat einen Podcast mit Molekularbiologe und Science Buster Martin Moder rund um die Gentechnik: "Die Optimierung des Menschen" (alexandria Podcast)
- Bibikova M, Golic M, Golic KG, Carroll D. (2002) Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases. Genetics Society of America, 61 pp. 1169–1175
- Boch J, Scholze H, Schornack S, Landgraf A, Hahn S, Kay S, Lehaya T, Nickstadt A, Bonas U. (2009) Breaking the Code of DNA Binding Specificity of TAL-Type III Effectors. Science Vol 326, p 1509. DOI: 10.1126/science.1178811
- Brown TA. (2011) Gentechnologie für Einsteiger (6. Aufl.) Springer Spektrum.
- Christian M, Cermak T, Doyle EL, Schmidt C, Zhang F, Hummel A, Bogdanove AJ, Voytas DF. (2010) Targeting DNA Double-Strand Breaks with TAL Effector Nucleases. Genetics Society of America, 186 pp. 757–761. DOI: 10.1534/genetics.110.120717
Folger KR, Wong EA, Wahl G, Capecchi MR. (1982), Patterns of Integration of DNA Microinjected into Cultured Mammalian Cells: Evidence for Homologous Recombination Between Injected Plasmid DNA Molecules. Molecular and cellular biology 2(11) p. 1372-1387. doi: 10.1128/mcb.2.11.1372
- Heberer B. (2015) Grüne Gentechnik. Hintergründe, Chancen und Risiken. Springer Spektrum. DOI 10.1007/978-3-658-09392-1
- Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A. (1987) Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product, Journal of Bacteriology. 169(12):5429-33. doi: 10.1128/jb.169.12.5429-5433.1987
- Gaj T, Gersbach CA, Barbas CF. (2013) ZFN, TALEN and CRISPR/Cas-based methods for genome engineering. Trends Biotechnol, 31(7) pp. 397–405. doi:10.1016/j.tibtech.2013.04.004
- Graw J. (2015) Genetik (6. Aufl.) Springer, 4. DOI 10.1007/978-3-662-44817-5
- Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. (2012), A programmable dual RNA-guided DNA endonuclease in adaptive bacterial immunity. Science 337(6096): 816–821. doi: 10.1126/science.1225829
- Kempken F, Kempken R. (2012) Gentechnik bei Pflanzen-Chancen und Risiken (4. Aufl.) Springer Spektrum, 7. DOI 10.1007/978-3-642-24818-4
- Kim Y, Cha J, Chandrasegaran S. (1996) Hybrid restriction enzymes: Zinc finger fusions to Fok I cleavage domain. Proc. Natl. Acad. Sci. USA Vol. 93, pp. 1156-1160. doi: 10.1073/pnas.93.3.1156
- Li L, Wu LP, Chandrasegaran S. (1992) Functional domains in Fok I restriction endonuclease. Proc. Natl. Acad. Sci. USA, Vol. 89, pp. 4275-4279. doi: 10.1073/pnas.89.10.4275
- Lloyd A, Plaiser CL, Carroll D, Drews GN. (2005) Targeted mutagenesis using zinc-finger nucleases in Arabidopsis. Proc. Natl. Acad. Sci. USA Vol. 102, pp. 2232–2237. doi: 10.1073/pnas.0409339102
- März S. (2017) Krebs ade – dank Gene Editing? Laborjournal. 09/2017 pp. 46-49
- Moscou MJ, Bogdanov AJ (2009) A simple cipher governs DNA Recognition by TAL Effectors. Science Vol 326 (5959), p 1501 . doi: 10.1126/science.1178817
- Mülhart C. (2013) Der Experimentator Molekularbiologie Genomics (7.Aufl.) Springer Spektrum. DOI 10.1007/978-3-642-34636-1
- Pattanayak V, Guilinger JP, Liu DR. (2014) Determining the Specificities of TALENs, Cas9, and other Genome-Editing Enzymes. Methods in Enzymology. Vol 546 pp. 47-78. DOI: 10.1016/B978-0-12-801185-0.00003-9
- Pfeiffer P, Thode S, Hancke J, Vielmetter W. (1994) Mechanisms of Overlap Formation in Nonhomologous DNA End Joining. Molecular and cellular biology 14 (2) p. 888-895. doi: 10.1128/mcb.14.2.888
- PubMed - NCBI (21.06.2020) Anzahl der wissenschaftlichen Publikationen (Stichworte „CRISPR/Cas9“ und „Genome-Editing“). https://pubmed.ncbi.nlm.nih.gov/
- Rembold M. (2017) Genauer zielen, besser schreiben. Laborjournal. 09/2017 pp. 35-41
- Schmidt O. (2017) Genetik und Molekularbiologie, Fritsche O. DOI 10.1007/978-3-662-50274-7
- Voytas DF, Gao C. (2014) Precision Genome Engineering and Agriculture: Opportunities and Regulatory Challenges. PLOS Biology, Vol 12. doi: 10.1371/journal.pbio.1001877
- Watson JD, Crick FH. (1953) Molecular Structure of Nucleic Acids; A Structure for Deoxyribose Nucleic Acid. Nature 171(4356):737-8. doi: 10.1038/171737a0

.jpg)


.png)